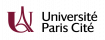Les objectifs fédérateurs de l’équipe sont les suivants :
- Tester des scénarios de croissance tumorale à l’aide de modèles simples
- Prédire l’évolution future de tumeurs
- Étudier et prédire l’effet de traitements sur les tumeurs
- Établir un lien entre la croissance tumorale étudiée à l’échelle micro (biologie) et à l’échelle macro (médecine)
- Développer des outils d’analyse d’images pour l’aide au diagnostic médical
Ces objectifs se déclinent selon trois axes de recherche :
1) Modélisation de données cliniques et biologiques des gliomes.
Ce thème de modélisation consiste à développer des modèles simples, avec un nombre de paramètres limité au minimum, et de les confronter à des données cliniques et/ou biologiques.
Formation d’agrégats de cellules tumorales (F98) sur des gels de PEG-PLL, à t=0 (en haut), t=6h (milieu) et t=12h (en bas) ; expériences (à gauche), simulations numériques (à droite). (d’après Adenis L, Gontran E, Deroulers C, Grammaticos B, Juchaux M, Seksek O, Badoual M. (2020), Experimental and modeling study of the formation of cell aggregates with differential substrate adhesion, PLoS One, 15, e0222371.)
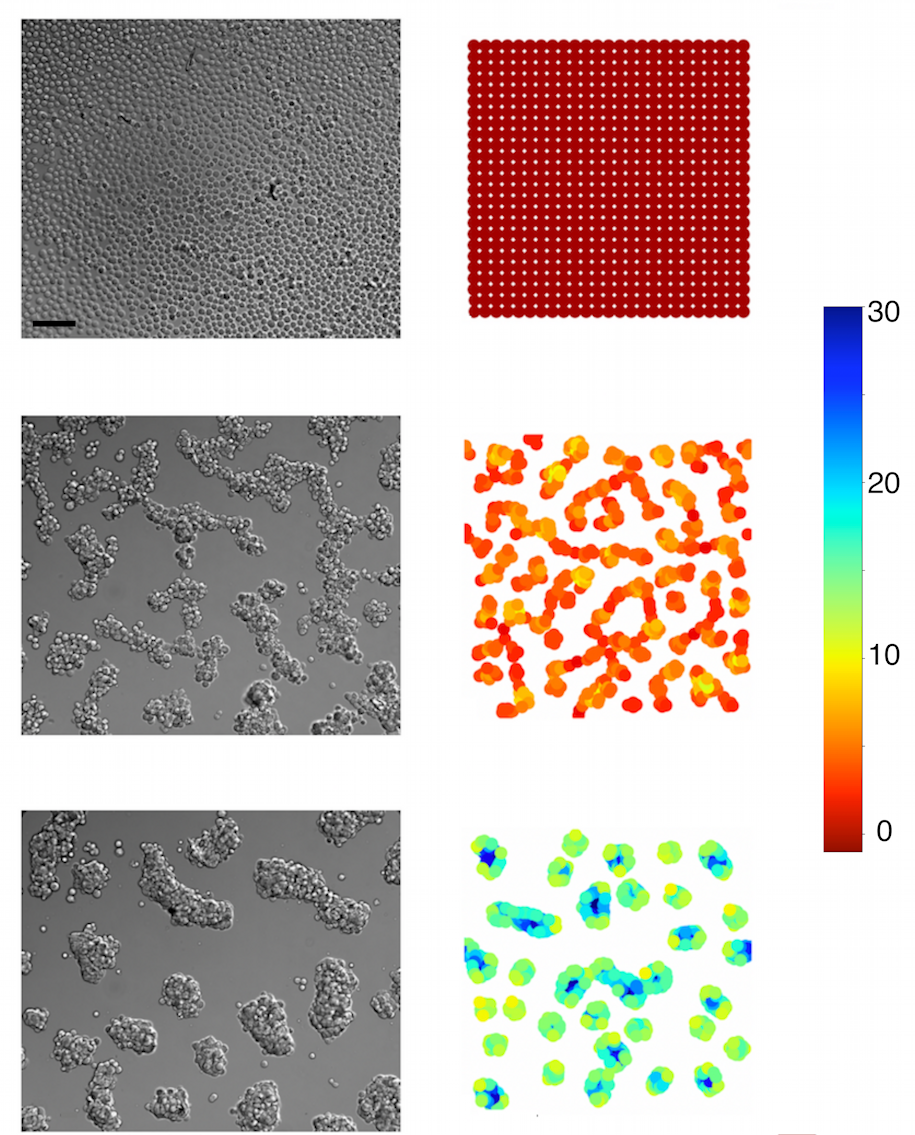
Exemple d’ajustement automatique de données cliniques du rayon tumoral (en mm, en ordonnée) en fonction du temps (en années, en abscisse) (d’après : Adenis L, Plaszczynski S, Grammaticos B, Pallud J, Badoual M, (2021), The Effect of Radiotherapy on Diffuse Low-Grade Gliomas Evolution: Confronting Theory with Clinical Data, J Pers Med., 11, 818).
Collaborations : Hôpital Sainte-Anne, Institut Curie, Laboratoire TIMC (Grenoble).
2) Physique statistique pour les gliomes.
Ce thème consiste à étudier les comportements collectifs d’assemblées de cellules, à partir de règles simples de déplacement cellulaire et d’interactions entre cellules et à l’aide d’outils de physique statistique (matière active).
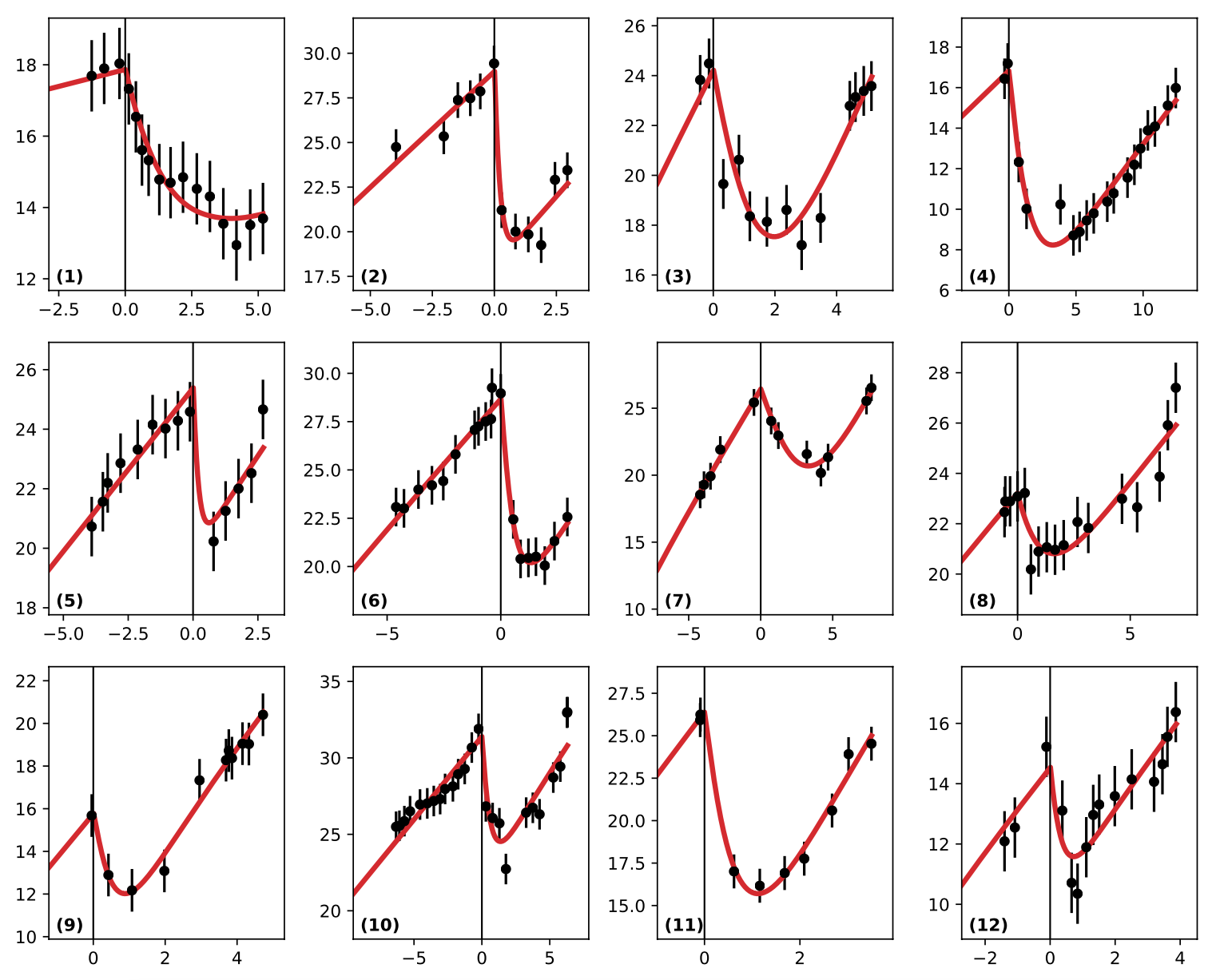
Formation d’agrégats dans le cas de la migration de cellules polarisées (d’après Nakamura G, Badoual M, Fabiani E and Deroulers D, (2021), Dispersal and organization of polarized cells: non-linear diffusion and cluster formation without adhesion, J. Stat. Mech. 093501).
3) Analyse big data de lames histologiques.
Ce thème consiste en de l’analyse de données, en vue d’alimenter à moyen terme les modèles mathématiques, d’images issues de la numérisation de lames histologiques. Cela passe par la mise au point de logiciels (objets de valorisation et/ou de publication) de manipulation et d’analyse d’images énormes (gigapixels).
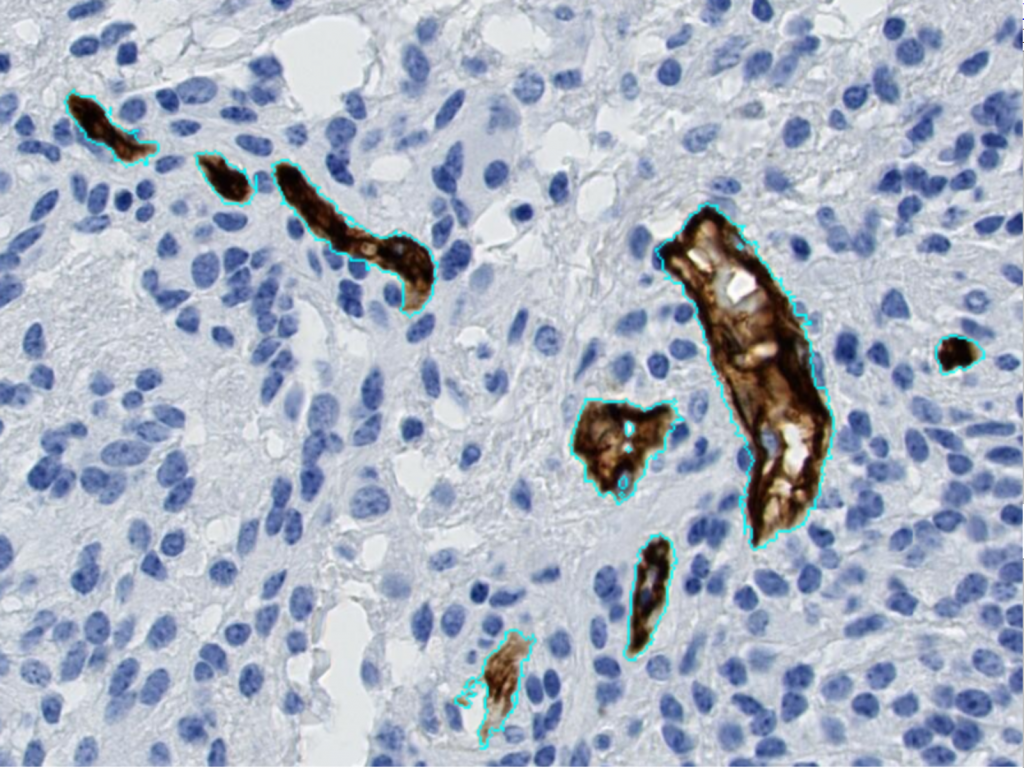
Exemple de segmentation automatique des vaisseaux sanguins (en marron, entourés en bleu cyan), sur un échantillon tissulaire provenant d’un gliome.
Collaborations: Hôpitaux Sainte-Anne, Necker, Kremlin-Bicêtre et Université de Caen.